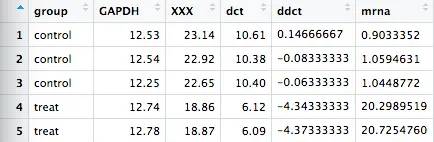
PCR数据要有三列,一列是组名,一列是内参基因的CT值,一列是目的基因的CT值,计算方法是-2∆∆Ct 法,实现一步出图用的是ggpubr,实现截断则是Y叔出手的ggbreak
- 列如下面这个表,定义为
PCR.csv,放桌面上:
| group | GAPDH | XXX |
|---|---|---|
| control | 12.53 | 23.14 |
| control | 12.54 | 22.92 |
| control | 12.25 | 22.65 |
| treat | 12.74 | 18.86 |
| treat | 12.78 | 18.87 |
| treat | 12.76 | 18.89 |
读取表格,并计算相对mRNA定量
PCR <- read_csv("~/Desktop/PCR.csv") #读表
PCR$dct=PCR$XXX-PCR$GAPDH ##目的基因Ct-内存基因Ct,即∆Ct
PCR$ddct=PCR$dct-mean(PCR$dct[1:3]) ##∆Ct-对照组Ct均值,即∆∆Ct
PCR$mrna=2^-PCR$ddct ##取-∆∆Ct的2次放,即-2^∆∆Ct
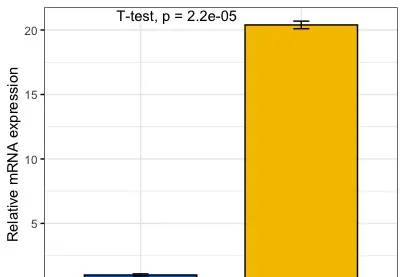
一步出图,ggpubr自带计算,均值、标准差、标准误,CI、P值都不在话下
library(ggpubr)
ggbarplot(PCR,
group ,
mrna ,
fill = group , # 按组填充颜色,当然如果喜爱单色,就用‘black’
palette = "jco", ## "npg", "aaas", "lancet"等主题任意选
add = "mean_sd",,xlab = F,ylab = Relative mRNA expression ,legend= none ,
ggtheme = theme_bw()#选一个自己喜爱的背景
)+stat_compare_means(method = t.test )
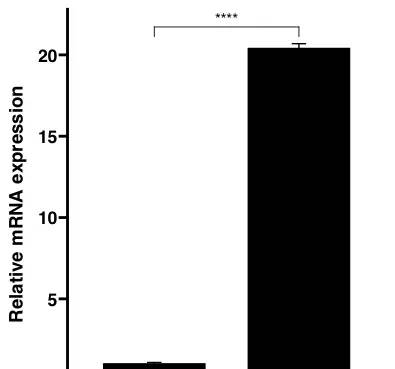
简单的优化一下,用上ggprism这个包,用上prism主题,冒充一下上用graphpad做的图
install.packages("ggprism")
library(ggprism)
ggbarplot(PCR,
group ,
mrna ,
fill = "black",
add = "mean_sd",
xlab = F,ylab = Relative mRNA expression ,legend= none ,
ggtheme = theme_prism()
)+stat_compare_means(aes(label = ..p.signif..), ## 改成星星
comparisons = list(c( control , treat )), ## 添加一下列表
method = t.test )
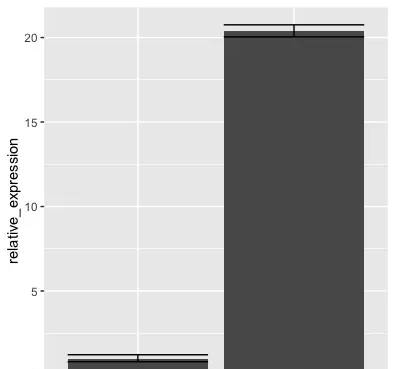
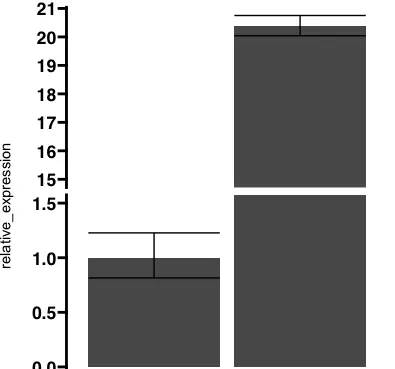
最后截断一下Y值
以前很少有包可以完美实现这个功能,我以前写过R做截断柱状图并加显著性统计可以实现,但Y叔出手写了个ggbreak包,完美的就解决了
install.packages("ggbreak")
library(ggbreak)
p<-ggbarplot(PCR,
group ,
mrna ,
fill = "black",
add = "mean_sd",
xlab = F,ylab = Relative mRNA expression ,legend= none ,
ggtheme = theme_prism()
)+stat_compare_means(aes(label = ..p.signif..),
comparisons = list(c( control , treat )),
method = t.test )
## 只要一句话就解决了,1.5是下轴,15是上轴,当然也可以三截断,定义宽度和高度,具体的自己可以研究
p+scale_y_break(c(1.5, 15) ,scales = c(1,10))

实则还有一个pcr的包也能简单实现,而且自动计算mRNA相对表达定量,而且对照组定量是1,更加科学,但是表格只是两列CT值,还要重新定义组,所以要先提取一下表格,处理一下数据。
install.packages("pcr")
library(pcr)
ct1<-PCR[,-c(1,4:6)]
group_var <- rep(c( control , treat ), each = 3)
result<-pcr_analyze(ct1,
group_var = group_var,
reference_gene = GAPDH ,
reference_group = control ,
method = delta_delta_ct )

实则pcr这个包也是可以直接出图的,还可以计算多基因,缺点是P值要另算,不能直接添加,所以要后期AI,出图直接加一句plot=T
library(pcr)
pcr_analyze(ct1,
group_var = group_var,
reference_gene = GAPDH ,
reference_group = control ,
method = delta_delta_ct ,plot=T)
也可以截断,还是ggbreak
pcr_analyze(ct1,
group_var = group_var,
reference_gene = GAPDH ,
reference_group = control ,
method = delta_delta_ct ,plot=T)+scale_y_break(c(1.5, 15) ,scales = c(1,10))+theme_prism()
© 版权声明
文章版权归作者所有,未经允许请勿转载。
相关文章
您必须登录才能参与评论!
立即登录









这个计算方式,如果不是两组,也就是不是“control”和“treat”,而是“group1”, “group2”, “group3”, “group4”,是不是不能这样计算了?因为我看这一步“PCR$ddct=PCR$dct-mean(PCR$dct[1:3]) ##∆Ct-对照组Ct均值,即∆∆Ct”中mean(PCR$dct[1:3])是一个定值